

Wir erhielten die Daten gleich im Anschluss an die Präsentationsvorstellung der verschiedenen Data Coaches von François Delavy vom SNF per USB-Stick. In diesem Abschnitt wird dieser Datensatz kurz erklärt.
Alle Forscher die am RNA & Disease Projekt teilnehmen, übermitteln dem SNSF jährlich ihre veröffentlichten Publikationen im Zusammenhang mit dem Projekt. Diese Daten enthalten die Forschungsgruppe und einige Informationen über die Publikation wie etwa den DOI, den Titel, die Namen der Autoren und so weiter. Mit diesen Daten wird anschliessend beim schweizerischen Nationalfonds gearbeitet, die Datenqualität ist dabei davon abhängig, wie gut die Mitglieder ihre Einträge vornehmen.
Zum Zeitpunkt als wir die Daten erhielten waren dies 54 Publikationen. Da das Projekt erst im Jahre 2014 startete, handelt es sich nur um Publikationen der letzten 3 Jahre. Ein NCCR Projekt kann aber bis zu 12 Jahre dauern, weshalb es sich zum Ende um sehr viele Daten handeln kann und eine Visualisierung umso mehr Nutzen schaffen kann. Gerade in den nächsten Jahren dürften sehr viele weitere Publikationen gemeldet werden, da es einige Zeit dauert und viel Aufwand für eine Publikation geleistet wird. Die Daten wurden anschliessend vom SNF (Im Falle unserer Daten von François Delavy) mittels Scopus API ergänzt. Bei dieser Ergänzung handelt es sich um Meta-Daten zu den Publikationen und zu den Autoren; namentlich die Zugehörigkeiten aller Autoren (das Forschungsinstitut) und das jeweilige Land. Bei dieser Suche wurden nicht alle Publikationen gefunden, vier Publikationen befinden sich deshalb nicht in der Visualisierung. Bei einer weiteren Publikation fehlten ausserdem die Meta-Daten, weshalb der Datensatz auf schliesslich auf 49 Publikationen reduziert wurde. Weiter ist die Datenqualität wiederum abhängig von den Angaben auf Scopus. Deshalb waren in den Namen der Forschungsinstitute zum Teil Fehler enthalten, wir haben dies gemeldet, und François Delavy hat die Daten für uns ausgebessert. In einem ersten Schritt haben wir die Daten dann so bereinigt, dass nur noch die Forschungsinstitute und die DOIs (Publikationen) enthalten waren. Diese Daten mussten wir anschliessend parsen, damit für jede Publikation jede Forschungsstelle einzeln angezeigt wird. Mit diesem Datensatz haben wir dann begonnen, unser Netzwerk zu erstellen.
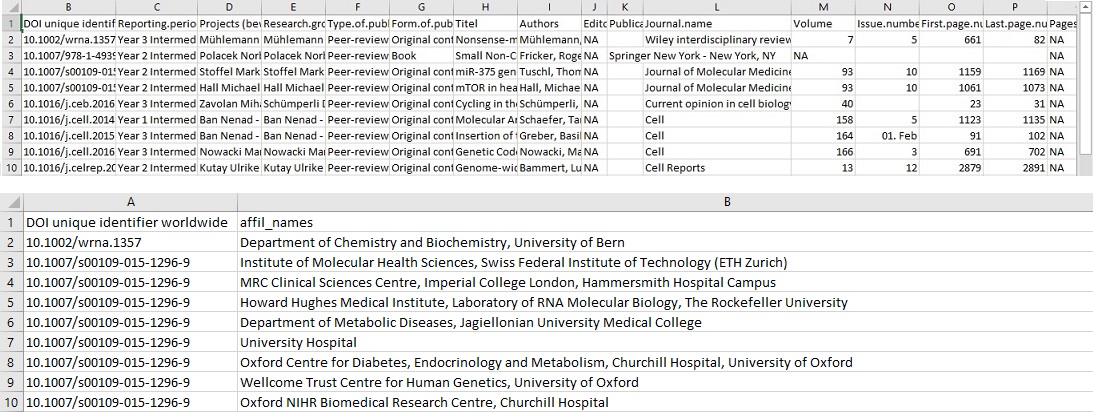
Abbildung 1: Zu sehen ist oben ein Ausschnitt aus dem ursprünglichen Datensatz der CSV-Datei. Unten ist ein Ausschnitt aus dem Datensatz, mit dem wir mit dem Visualisieren begonnen haben. DOI ist die Publikation, affil_names ist der Name der Forschunggstelle. Gut zu sehen ist auf der unteren Abbildung, wie wir die Daten geparst haben. Die Einträge 3 bis 10 waren im ursprünglichen Datensatz auf einer Zeile, da sie zur selben Publikation gehören.
Das Problem in diesem Datensatz war, dass einige Forschungsinstitute an mehreren Publikationen beteiligt waren und es dadurch Duplikate bei den Nodes gab. In einem weiteren Schritt wurde deshalb ein Datensatz nur mit den Forschungsinstituten erstellt und die Duplikate wurden entfernt. Diese Datei wurde in ein JSON-File konvertiert, direkt als Variable in unsere Visualisierung aufgenommen (nicht als externes JSON-File) und so hatten wir unsere Nodes.

Abbildung 2: Jede "id" entspricht einer Node.
Um ein Netzwerk zu erstellen ist normalerweise ein Datensatz mit den Einträgen «source» und «target» erforderlich. Um dieses Problem zu umgehen und unsere Verbindungen direkt aus dem File mit den Forschungsinstituten und Publikation zu generieren, mussten wir wieder auf unser erstes File zurück greifen und einen for loop erstellen:

Abbildung 3: Oben ein Auszug der Daten, die für die Links benötigt wurden. "id" ist das Forschungsinstitut und "group" die Publikation.
Dieser loop geht das File durch und erstellt überall wo die gleiche Publikation eingetragen ist, einen Link mit einer «source» und einem «target». Dadurch konnten wir unsere Links erstellen und hatten somit die Basis für unser Netzwerk. Im Verlauf des Projektes kamen dann zu diesen Daten bei jeder Forschungsstelle noch das Land dazu, damit alle Forschungsinstitute aus einem Land in der Visualisierung dieselbe Farbe haben und in der Visualisierung als Legende beschrieben werden konnten. Diese Daten befanden sich auch im originalen Datensatz, wir mussten sie auch parsen, genau gleich wie wir dies zuvor für die Forschunggstellen gemacht haben. Diese Daten wurden wiederum als Variable in unsere Datei aufgenommen. In einem letzten Schritt haben wir dann noch einen zusätzlichen Datensatz erstellt, bei dem für jede Forschungsstelle alle Publikationen angezeigt werden. Dies war nötig um die DOIs anzuzeigen wenn auf die Node geklickt wird.

Abbildung 3: Ausschnitt aus dem JSON-File der letzten Daten, die wir für die Visualisierung verwendet haben. Für jede Forschungsstelle ("name") werden die zugehörigen Publikationen ("DOI") angezeigt.
Die App zeigt die verschiedenen am Projekt beteiligten Forschungsinstitute als Nodes. Durch die verschiedenen Farben wird schneller ersichtlich, welche Nodes aus der Schweiz sind und dadurch mögliche Mitglieder des Projekts sind. In der Legende links wird jeweils für jede Farbe das Ursprungsland angezeigt. Je mehr eine Forschungsstelle publiziert hat (an Publikationen beteiligt war), desto grösser wird die Node. Durch den mouseover-Effekt wird bei der Node der Name angezeigt, wenn man darüberfährt. Durch einen Klick auf eine Node werden ein Alert mit dem Namen der Forschungsstelle sowie die DOIs angezeigt. Mit den Buttons oberhalb des Netzwerks können die drei verschiedenen Netzwerke angeschaut werden. Für den SNF ist die Entwicklung sehr wichtig, weshalb nicht jedes Jahr einzeln, sondern die Kumulation im Vordergrund standen. Das Netzwerk Jahr 1 ist also aus dem Jahr 2014, das Netzwerk Jahre 1-2 aus den Jahren 2014 und 2015 und das Netzwerk Jahre 1-3 schliesslich für die Jahre 2014-2016. Die App ist ausserdem responsive, passt sich also der Bildschirmgrösse automatisch an.
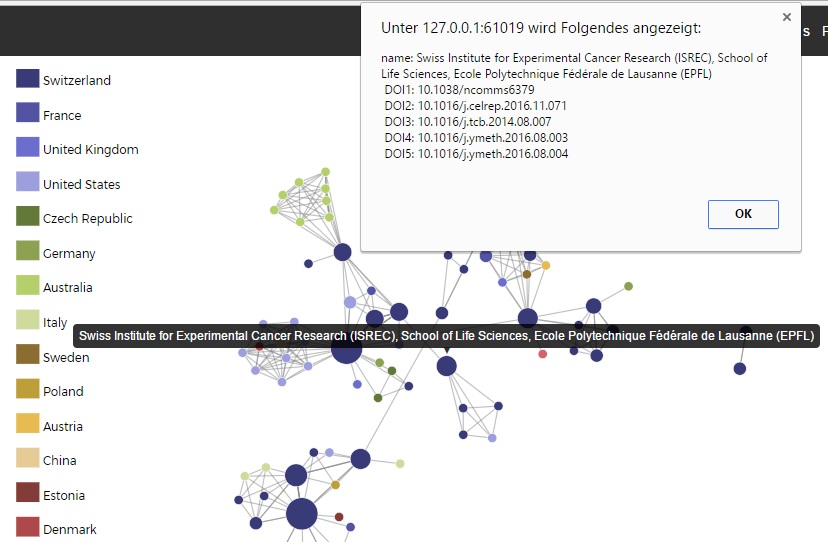
Abbildung 4: Screenshot aus der Visualisierung, Netzwerk Jahre 1-3. Auf diesem Bild sind der Mouseover-Effekt sowie der Alert, der bei einem Klick erscheint, zu sehen. Durch den Alert werden die verschiedenen DOIs sichtbar, ausserdem ist so die Anzahl der DOIs pro Forschungsstelle genau erkennbar.
Durch das Anklicken der Buttons oberhalb der Visualisierung ist die Entwicklung über die Zeit gut erkennbar, also wie die einzelnen Nodes durch neue Publikationen grösser geworden sind und auch wie neue Nodes dazugekommen sind. Der ursprüngliche gewünschte Output von François Delavy wurde mit dem Netzwerk erreicht. Es ist sehr übersichtlich, welche Forschungsinstitute im Projekt "RNA and Disease" durch Co-Publikationen zusammengearbeitet haben. Im "Netzwerk Jahre 1-3" ist beispielsweise sehr gut erkennbar, dass es insgesamt drei Institute gibt (2 Mal von der ETH Zürich, 1 Mal von der Universität Bern), die alle ihr "eigenes Netzwerk" haben, mit dem sie zusammengearbeitet haben in Abbildung 5 gut zu erkennen). In der Legende kann zudem bei Interesse nachgeschaut werden, um welches Land es sich handelt und wie viele Länder bereits beteiligt waren. Da sehr viele der Nodes dunkelblau sind, ist gut erkennbar, dass sehr viele Institute aus der Schweiz beteiligt sind. Durch die Visualisierung wird ausserdem schnell offensichtlich, welche Forschungsinstitute bis jetzt am meisten publiziert haben (oder genauer, dies dem SNF bereits gemeldet haben). Es ist augenblicklich zu sehen, dass die Nodes des «Department of Chemistry and Biochemistry, University of Bern» sowie das «Institute of Molecular Biology and Biophysics, Department of Biology, ETH Zurich» am grössten sind und folglich am meisten publiziert haben.
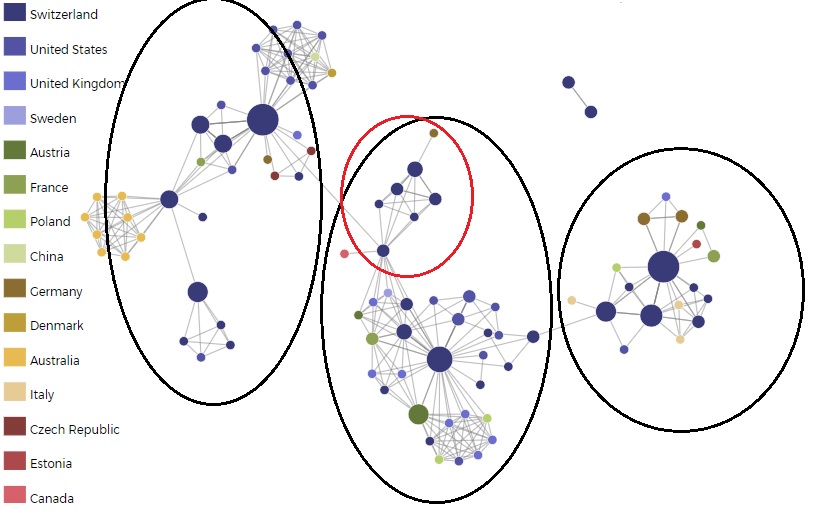
Abbildung 5: Schwarz eingekreist die drei aktivsten Forschungsinstitute mit ihrem "eigenen" Netzwerk. Mit Vorsicht muss bei die rot eingekreiste Forschungsstelle betrachtet werden, sie hat als einzige mit zwei der grossen Forschungsinstitute publiziert.